Projektowanie leków - Drug design
|
|
Projektowanie leków , często określane jako racjonalne projektowanie leków lub po prostu racjonalne projektowanie , jest wynalazczym procesem znajdowania nowych leków w oparciu o wiedzę o celu biologicznym . Lek jest najczęściej organiczną małą cząsteczką, która aktywuje lub hamuje funkcję biocząsteczki, takiej jak białko , co z kolei daje pacjentowi korzyść terapeutyczną . W najbardziej podstawowym sensie projektowanie leków obejmuje projektowanie cząsteczek, które są komplementarne pod względem kształtu i ładunku do celu biomolekularnego, z którym oddziałują, a zatem będą się z nim wiązać. Projektowanie leków często, ale niekoniecznie, opiera się na technikach modelowania komputerowego . Ten rodzaj modelowania jest czasami określany jako komputerowe wspomaganie projektowania leków . Wreszcie projektowanie leków, które opiera się na wiedzy o trójwymiarowej strukturze celu biomolekularnego, znane jest jako projektowanie leków oparte na strukturze . Oprócz małych cząsteczek, opracowano również biofarmaceutyki, w tym peptydy, a zwłaszcza przeciwciała terapeutyczne, które stanowią coraz ważniejszą klasę leków i metod obliczeniowych poprawiających powinowactwo, selektywność i stabilność tych środków terapeutycznych opartych na białkach.
Wyrażenie „projektowanie leków” jest do pewnego stopnia mylące . Bardziej dokładnym terminem jest projektowanie ligandów (tj. projektowanie cząsteczki, która będzie ściśle wiązać się ze swoim celem). Chociaż techniki projektowania do przewidywania powinowactwa wiązania są dość skuteczne, istnieje wiele innych właściwości, takich jak biodostępność , metaboliczny okres półtrwania , skutki uboczne itp., które najpierw należy zoptymalizować, zanim ligand stanie się bezpiecznym i skutecznym lekiem. Te inne cechy są często trudne do przewidzenia za pomocą racjonalnych technik projektowania. Niemniej jednak, z powodu dużej szybkości ścierania, zwłaszcza podczas faz klinicznych w rozwoju leków więcej uwagi skupia jest na wczesnym etapie procesu opracowywania leków o wyborze leków-kandydatów, których dane fizykochemiczne właściwości przewiduje spowodować mniej komplikacji podczas rozwoju, a zatem bardziej prawdopodobnie prowadzić do zatwierdzony, wprowadzony do obrotu lek. Co więcej, eksperymenty in vitro uzupełnione metodami obliczeniowymi są coraz częściej wykorzystywane we wczesnym odkrywaniu leków, aby wybrać związki o korzystniejszych profilach ADME (wchłanianie, dystrybucja, metabolizm i wydalanie) i toksykologicznych .
Cele narkotykowe
Cel biomolekularny (najczęściej białko lub kwas nukleinowy ) jest kluczową cząsteczką zaangażowaną w konkretny szlak metaboliczny lub sygnałowy , który jest związany z określonym stanem chorobowym lub patologią lub z zakaźnością lub przeżyciem patogenu drobnoustrojowego . Potencjalne cele leków niekoniecznie są przyczyną choroby, ale z definicji muszą być modyfikujące chorobę. W niektórych przypadkach małe cząsteczki będą zaprojektowane tak, aby wzmacniać lub hamować funkcję docelową w określonym szlaku modyfikującym chorobę. Zaprojektowane zostaną małe cząsteczki (na przykład agoniści , antagoniści , odwrotni agoniści lub modulatory receptora ; aktywatory lub inhibitory enzymów ; albo środki otwierające lub blokujące kanały jonowe ), które są komplementarne do miejsca wiązania celu. Małe cząsteczki (leki) można zaprojektować tak, aby nie wpływały na żadne inne ważne cząsteczki „poza-docelowe” (często określane jako przeciw -docelowe), ponieważ interakcje leków z cząsteczkami niedocelowymi mogą prowadzić do niepożądanych skutków ubocznych . Ze względu na podobieństwa w miejscach wiązania, blisko spokrewnione cele zidentyfikowane przez homologię sekwencji mają największą szansę na reaktywność krzyżową, a zatem najwyższy potencjał skutków ubocznych.
Najczęściej leki są małymi cząsteczkami organicznymi wytwarzanymi w wyniku syntezy chemicznej, ale leki na bazie biopolimerów (znane również jako biofarmaceutyki ) wytwarzane w procesach biologicznych stają się coraz bardziej powszechne. Ponadto technologie wyciszania genów oparte na mRNA mogą mieć zastosowania terapeutyczne.
Racjonalne odkrywanie leków
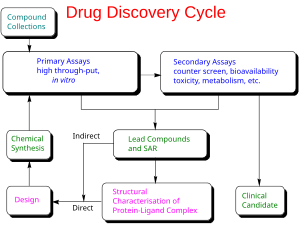
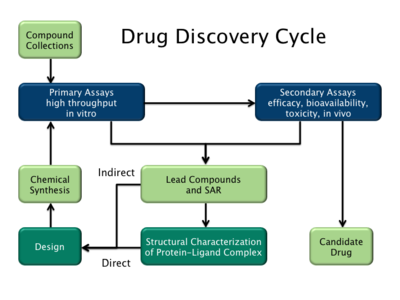
W przeciwieństwie do tradycyjnych metod odkrywania leków (znanych jako forward pharmacology ), które polegają na testowaniu metodą prób i błędów substancji chemicznych na hodowanych komórkach lub zwierzętach i dopasowywaniu widocznych efektów do leczenia, racjonalne projektowanie leków (zwane również odwrotną farmakologią ) zaczyna się od hipotezy, że modulacja określonego celu biologicznego może mieć wartość terapeutyczną. Aby biocząsteczka mogła zostać wybrana jako cel leku, wymagane są dwie podstawowe informacje. Pierwszy to dowód na to, że modulacja celu będzie modyfikacją choroby. Ta wiedza może pochodzić na przykład z badań powiązań chorobowych, które wykazują związek między mutacjami w celu biologicznym a pewnymi stanami chorobowymi. Po drugie, cel jest „ podatny na narkotyki ”. Oznacza to, że jest zdolny do wiązania się z małą cząsteczką i że jego aktywność może być modulowana przez małą cząsteczkę.
Gdy odpowiedni cel został zidentyfikowany, cel zwykle sklonowano i produkowane i oczyszcza . Oczyszczone białko jest następnie wykorzystywane do ustalenia testu przesiewowego . Dodatkowo można określić trójwymiarową strukturę celu.
Poszukiwanie małych cząsteczek wiążących się z celem rozpoczyna się od przeszukiwania bibliotek potencjalnych związków leczniczych. Można to zrobić za pomocą testu przesiewowego („mokry ekran”). Ponadto, jeśli dostępna jest struktura celu, można przeprowadzić wirtualny przesiew kandydatów na leki. Idealnie, kandydujące związki lecznicze powinny być „ lekopodobne ”, to znaczy powinny mieć właściwości, które, jak się przewiduje, prowadzą do biodostępności doustnej , odpowiedniej stabilności chemicznej i metabolicznej oraz minimalnych skutków toksycznych. Dostępnych jest kilka metod oceny podobieństwa do leków, takich jak reguła pięciu Lipińskiego oraz szereg metod oceny, takich jak skuteczność lipofilna . W literaturze naukowej zaproponowano również kilka metod przewidywania metabolizmu leków.
Ze względu na dużą liczbę właściwości leków, które muszą być jednocześnie optymalizowane podczas procesu projektowania, czasami stosuje się wielocelowe techniki optymalizacji . Wreszcie, z powodu ograniczeń obecnych metod przewidywania aktywności, projektowanie leków nadal w dużym stopniu opiera się na zbiegu okoliczności i ograniczonej racjonalności .
Wspomagane komputerowo projektowanie leków
Najbardziej podstawowym celem w projektowaniu leków jest przewidzenie, czy dana cząsteczka będzie wiązać się z celem, a jeśli tak, jak silnie. Do oceny siły oddziaływania międzycząsteczkowego między małą cząsteczką a jej biologicznym celem najczęściej stosuje się mechanikę molekularną lub dynamikę molekularną . Metody te są również wykorzystywane do przewidywania konformacji małej cząsteczki i modelowania zmian konformacyjnych w celu, które mogą wystąpić, gdy mała cząsteczka się z nim zwiąże. Metody półempiryczne , metody chemii kwantowej ab initio lub teoria funkcjonału gęstości są często stosowane w celu zapewnienia zoptymalizowanych parametrów do obliczeń mechaniki molekularnej, a także oszacowania właściwości elektronowych (potencjał elektrostatyczny, polaryzowalność itp.) potencjalnego leku, który będzie wpływać na powinowactwo wiązania.
Do półilościowej prognozy powinowactwa wiązania można również zastosować metody mechaniki molekularnej. Ponadto funkcja punktacji opartej na wiedzy może być wykorzystana do zapewnienia wiążących oszacowań powinowactwa. Metody te wykorzystują regresję liniową , uczenie maszynowe , sieci neuronowe lub inne techniki statystyczne w celu uzyskania predykcyjnych równań powinowactwa wiązania poprzez dopasowanie eksperymentalnych powinowactw do wyprowadzonych obliczeniowo energii interakcji między małą cząsteczką a celem.
W idealnej sytuacji metoda obliczeniowa będzie w stanie przewidzieć powinowactwo przed zsyntetyzowaniem związku, a zatem teoretycznie wystarczy zsyntetyzować tylko jeden związek, co oszczędzi ogromną ilość czasu i kosztów. Rzeczywistość jest taka, że obecne metody obliczeniowe są niedoskonałe i dostarczają w najlepszym razie tylko jakościowo dokładne szacunki powinowactwa. W praktyce odkrycie optymalnego leku wymaga kilku iteracji projektowania, syntezy i testowania. Metody obliczeniowe przyspieszyły odkrywanie poprzez zmniejszenie liczby wymaganych iteracji i często dostarczały nowych struktur.
Projektowanie leków za pomocą komputerów może być wykorzystywane na każdym z następujących etapów odkrywania leków:
- identyfikacja trafień za pomocą wirtualnego skriningu (projektowanie oparte na strukturze lub ligandach)
- optymalizacja powinowactwa i selektywności hit-to-lead (projektowanie w oparciu o strukturę, QSAR itp.)
- optymalizacja ołowiu innych właściwości farmaceutycznych przy zachowaniu powinowactwa
W celu przezwyciężenia niewystarczającego przewidywania powinowactwa wiązania obliczonego na podstawie ostatnich funkcji zliczania, do analizy wykorzystuje się oddziaływanie białko-ligand i informacje o strukturze 3D związku. W przypadku projektowania leków opartych na strukturze opracowano kilka analiz po przesiewowych skupiających się na interakcji białko-ligand w celu poprawy wzbogacania i skutecznego wyszukiwania potencjalnych kandydatów:
- Punktacja konsensusu
- Wybór kandydatów poprzez głosowanie wielu funkcji punktacji
- Może utracić związek między strukturą informacji białko-ligand a kryterium punktacji
- Analiza skupień
- Reprezentuj i skupiaj kandydatów zgodnie z informacjami 3D białko-ligand
- Potrzebuje znaczącej reprezentacji interakcji białko-ligand.
Rodzaje
Istnieją dwa główne rodzaje projektowania leków. Pierwszy jest nazywany ligandem opartym projektowaniu leków , a drugi, na podstawie struktury Drug Design .
Oparte na ligandach
Projektowanie leków oparte na ligandach (lub projektowanie leków pośrednich ) opiera się na wiedzy o innych molekułach, które wiążą się z biologicznym celem będącym przedmiotem zainteresowania. Te inne cząsteczki można wykorzystać do uzyskania modelu farmakoforu, który określa minimalne niezbędne cechy strukturalne, jakie musi posiadać cząsteczka, aby związać się z celem. Innymi słowy, model celu biologicznego można zbudować w oparciu o wiedzę o tym, co się z nim wiąże, a ten model z kolei można wykorzystać do projektowania nowych jednostek molekularnych, które oddziałują z celem. Alternatywnie, można wyprowadzić ilościową zależność struktura-aktywność (QSAR), w której można wyprowadzić korelację między obliczonymi właściwościami cząsteczek a ich eksperymentalnie określoną aktywnością biologiczną . Te zależności QSAR z kolei można wykorzystać do przewidywania aktywności nowych analogów.
Oparte na strukturze
Projektowanie leków oparte na strukturze (lub bezpośrednie projektowanie leków ) opiera się na znajomości trójwymiarowej struktury celu biologicznego uzyskanej za pomocą metod takich jak krystalografia rentgenowska lub spektroskopia NMR . Jeśli eksperymentalna struktura celu jest niedostępna, możliwe jest stworzenie modelu homologii celu w oparciu o eksperymentalną strukturę pokrewnego białka. Wykorzystując strukturę celu biologicznego, kandydaci na leki, co do których przewiduje się, że będą się z nim wiązać z wysokim powinowactwem i selektywnością, można zaprojektować za pomocą interaktywnej grafiki i intuicji chemika medycznego . Alternatywnie można zastosować różne zautomatyzowane procedury obliczeniowe do sugerowania nowych kandydatów na leki.
Obecne metody projektowania leków w oparciu o strukturę można z grubsza podzielić na trzy główne kategorie. Pierwsza metoda to identyfikacja nowych ligandów dla danego receptora poprzez przeszukiwanie dużych baz danych struktur 3D małych cząsteczek w celu znalezienia tych, które pasują do kieszeni wiążącej receptora za pomocą szybkich programów dokowania aproksymującego . Ta metoda jest znana jako wirtualne badanie przesiewowe . Druga kategoria to projektowanie nowych ligandów de novo. W tej metodzie cząsteczki ligandu są budowane w ramach ograniczeń kieszeni wiążącej przez składanie małych kawałków w sposób stopniowy. Te kawałki mogą być pojedynczymi atomami lub fragmentami cząsteczek. Kluczową zaletą takiej metody jest możliwość zaproponowania nowych struktur, nie zawartych w żadnej bazie danych. Trzecią metodą jest optymalizacja znanych ligandów poprzez ocenę proponowanych analogów we wnęce wiążącej.
Wiążąca identyfikacja strony
Identyfikacja miejsca wiązania to pierwszy krok w projektowaniu opartym na strukturze. Jeśli struktura celu lub wystarczająco podobnego homologu jest określana w obecności związanego ligandu, wówczas ligand powinien być obserwowalny w strukturze, w którym to przypadku lokalizacja miejsca wiązania jest trywialna. Mogą jednak istnieć niezajęte allosteryczne miejsca wiązania, które mogą być interesujące. Ponadto może się zdarzyć, że dostępne są tylko struktury apoproteiny (białko bez liganda), a wiarygodna identyfikacja niezajętych miejsc, które mogą wiązać ligandy z wysokim powinowactwem, nie jest trywialna. W skrócie, identyfikacja miejsca wiązania zwykle opiera się na identyfikacji wklęsłych powierzchni białka, które mogą pomieścić cząsteczki wielkości leku, które również posiadają odpowiednie „gorące punkty” ( powierzchnie hydrofobowe , miejsca wiązania wodorowego itp.), które napędzają wiązanie liganda.
Funkcje punktacji
Structure-based drug design próbuje wykorzystać strukturę białek jako podstawę do projektowania nowych ligandów poprzez zastosowanie zasad rozpoznawania molekularnego . Selektywne wiązanie o wysokim powinowactwie z celem jest ogólnie pożądane, ponieważ prowadzi do bardziej skutecznych leków z mniejszą liczbą skutków ubocznych. Tak więc, jedną z najważniejszych zasad projektowania lub otrzymywania potencjalnych nowych ligandów jest przewidywanie powinowactwa wiązania określonego liganda do jego celu (i znanych antycelów ) i wykorzystanie przewidywanego powinowactwa jako kryterium selekcji.
Böhm opracował jedną wczesną, empiryczną funkcję punktacji ogólnego przeznaczenia, opisującą energię wiązania ligandów z receptorami. Ta empiryczna funkcja scoringowa przybrała postać:
gdzie:
- ΔG 0 – empirycznie wyprowadzony offset, który częściowo odpowiada całkowitej utracie entropii translacyjnej i rotacyjnej liganda po związaniu.
- ΔG hb – udział wiązania wodorowego
- ΔG jonowy – udział oddziaływań jonowych
- ΔG warga – udział oddziaływań lipofilowych, gdzie | Alipo | to powierzchnia kontaktu lipofilowego liganda z receptorem
- ΔG rot – kara entropii spowodowana zamrożeniem elementu obrotowego w wiązaniu ligandowym podczas wiązania
Bardziej ogólne termodynamiczne równanie „główne” jest następujące:
gdzie:
- desolwatacja – kara entalpiczna za usunięcie liganda z rozpuszczalnika
- ruch – entropia kara za zmniejszenie stopni swobody, gdy ligand wiąże się z jego receptorem
- konfiguracja – energia odkształcenia konformacyjnego potrzebna do wprowadzenia liganda w jego „aktywną” konformację
- interakcja – zysk entalpiczny dla „rozdzielenia” liganda z jego receptorem
Podstawową ideą jest to, że ogólna swobodna energia wiązania może zostać rozłożona na niezależne składniki, o których wiadomo, że są ważne dla procesu wiązania. Każdy składnik odzwierciedla pewien rodzaj zmiany energii swobodnej podczas procesu wiązania między ligandem a jego docelowym receptorem. Równanie główne to liniowa kombinacja tych składników. Zgodnie z równaniem energii swobodnej Gibbsa zbudowano zależność między stałą równowagi dysocjacji, K d , a składnikami energii swobodnej.
Do oszacowania każdego ze składników równania głównego wykorzystywane są różne metody obliczeniowe. Na przykład zmianę pola powierzchni polarnej po związaniu liganda można wykorzystać do oszacowania energii desolwatacji. Liczba obrotowych wiązań zamrożonych po związaniu liganda jest proporcjonalna do czasu ruchu. Energię konfiguracji lub odkształcenia można oszacować za pomocą obliczeń mechaniki molekularnej . Wreszcie energię interakcji można oszacować za pomocą metod, takich jak zmiana powierzchni niepolarnej, statystycznie wyprowadzone potencjały średniej siły , liczba utworzonych wiązań wodorowych itp. W praktyce elementy równania głównego są dopasowane do danych eksperymentalnych przy użyciu wielu regresja liniowa. Można to zrobić za pomocą zróżnicowanego zestawu treningowego obejmującego wiele rodzajów ligandów i receptorów, aby uzyskać mniej dokładny, ale bardziej ogólny „globalny” model lub bardziej ograniczony zestaw ligandów i receptorów, aby uzyskać dokładniejszy, ale mniej ogólny model „lokalny”.
Przykłady
Szczególnym przykładem racjonalnego projektowania leków jest wykorzystanie trójwymiarowej informacji o biocząsteczkach uzyskanych z takich technik jak krystalografia rentgenowska i spektroskopia NMR. Wspomagane komputerowo projektowanie leków w szczególności staje się znacznie bardziej przystępne, gdy istnieje struktura docelowego białka o wysokiej rozdzielczości związana z silnym ligandem. Takie podejście do odkrywania leków jest czasami określane jako projektowanie leków oparte na strukturze. Pierwszym jednoznacznym przykładem zastosowania projektowania leku opartego na strukturze prowadzącego do uzyskania zatwierdzonego leku jest inhibitor anhydrazy węglanowej dorzolamid , który został zatwierdzony w 1995 roku.
Inną ważną badania miało miejsce w racjonalnym projektowaniu leków jest imatynib , A kinazy tyrozynowej, inhibitory przeznaczone specyficznie dla BCR-Abl białka fuzyjnego, który jest charakterystyczny dla chromosomem Philadelphia dodatnich białaczki ( przewlekłej białaczki szpikowej , a czasami ostrej białaczki szpikowej ). Imatinib zasadniczo różni się od poprzednich leków na raka , ponieważ większość środków chemioterapii po prostu celuje w szybko dzielące się komórki, nie różnicując komórek rakowych i innych tkanek.
Dodatkowe przykłady obejmują:
- Wiele atypowych leków przeciwpsychotycznych
- Cymetydyna , prototypowym H 2 antagonistą receptora , z których zostały opracowane później członków klasy
- Selektywne inhibitory COX-2, inhibitorami NLPZ
- Enfuwirtyd , peptydowy inhibitor wnikania wirusa HIV
- Niebenzodiazepiny, takie jak zolpidem i zopiklon
- Raltegrawir , inhibitor integrazy HIV
- SSRI (selektywne inhibitory wychwytu zwrotnego serotoniny), klasa leków przeciwdepresyjnych
- Zanamiwir , lek przeciwwirusowy
Studium przypadku
- Antagoniści 5-HT3
- Agoniści receptora acetylocholiny
- Antagoniści receptora angiotensyny
- Inhibitory kinazy tyrozynowej Bcr-Abl
- Antagoniści receptora kannabinoidowego
- Antagoniści receptora CCR5
- Inhibitory cyklooksygenazy 2
- Inhibitory dipeptydylopeptydazy-4
- Inhibitory proteazy HIV
- Antagoniści receptora NK1
- Nienukleozydowe inhibitory odwrotnej transkryptazy
- Nukleozydowe i nukleotydowe inhibitory odwrotnej transkryptazy
- Inhibitory PDE5
- Inhibitory pompy protonowej
- Inhibitory reniny
- Tryptany
- Antagoniści TRPV1
- Inhibitory c-Met
Krytyka
Argumentowano, że wysoce sztywna i skoncentrowana natura racjonalnego projektowania leków tłumi nieoczekiwane odkrycie leków.
Zobacz też
Bibliografia
Zewnętrzne linki
- Drug+Design w Narodowej Bibliotece Medycznej USA Medical Subject Headings (MeSH)



![{\ Displaystyle {\ zacząć {tablica} {lll} \ Delta G_ {\ tekst {powiązanie}} = - RT \ ln K_ {\ tekst {d}} \ \ [1.3ex] K_ {\ tekst {d}} = {\dfrac {[{\text{Ligand}}][{\text{Receptor}}]}{[{\text{Złożony}}]}}\\[1.3ex]\Delta G_{\text{bind} }=\Delta G_{\text{desolwatacja}}+\Delta G_{\text{ruch}}+\Delta G_{\text{konfiguracja}}+\Delta G_{\text{interakcja}}\end{tablica} }}](https://wikimedia.org/api/rest_v1/media/math/render/svg/ba49ddd9dec7415d129787213744ca1afcd2d021)