Filogenetyczne metody porównawcze - Phylogenetic comparative methods
Filogenetyczne metody porównawcze ( PCM ) wykorzystują informacje o historycznych związkach rodów ( filogenezy ) do testowania hipotez ewolucyjnych. Metoda porównawcza ma długą historię w biologii ewolucyjnej; rzeczywiście, Karol Darwin wykorzystał różnice i podobieństwa między gatunkami jako główne źródło dowodów w O powstawaniu gatunków . Jednak fakt, że blisko spokrewnione rody mają wiele cech i kombinacji cech w wyniku procesu pochodzenia z modyfikacją oznacza, że rody nie są niezależne. Ta realizacja zainspirowała rozwój wyraźnie filogenetycznych metod porównawczych. Początkowo metody te zostały opracowane przede wszystkim w celu kontroli historii filogenetycznej podczas testowania adaptacji ; jednak w ostatnich latach użycie tego terminu rozszerzyło się na wszelkie użycie filogenezy w testach statystycznych. Chociaż większość badań wykorzystujących PCM koncentruje się na istniejących organizmach, wiele metod można również zastosować do wymarłych taksonów i mogą uwzględniać informacje z zapisu kopalnego .
PCM można ogólnie podzielić na dwa rodzaje podejść: te, które wnioskować o historii ewolucyjnej jakiegoś charakteru ( fenotypowego lub genetycznego ) w całej filogenezie i te, które wnioskować o samym procesie ewolucyjnego rozgałęzienia ( wskaźniki dywersyfikacji ), chociaż istnieją pewne podejścia, które oba jednocześnie. Zazwyczaj drzewo, które jest używane w połączeniu z PCM, zostało oszacowane niezależnie (patrz filogenetyka obliczeniowa ), tak że zakłada się, że znane są zarówno relacje między liniami, jak i długość gałęzi, które je oddzielają.
Aplikacje
Filogenetyczne podejścia porównawcze mogą uzupełniać inne sposoby badania adaptacji, takie jak badanie populacji naturalnych, badania eksperymentalne i modele matematyczne. Porównania międzygatunkowe pozwalają naukowcom ocenić ogólność zjawisk ewolucyjnych poprzez rozważenie niezależnych zdarzeń ewolucyjnych. Takie podejście jest szczególnie przydatne, gdy zmienność w obrębie gatunku jest niewielka lub nie ma jej wcale. A ponieważ można ich używać do jawnego modelowania procesów ewolucyjnych zachodzących w bardzo długich okresach czasu, mogą zapewnić wgląd w zagadnienia makroewolucyjne, niegdyś wyłączną domenę paleontologii .

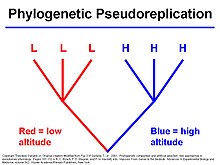
Filogenetyczne metody porównawcze są powszechnie stosowane do takich pytań jak:
- Jakie jest nachylenie o allometrycznej relacji skalowania?
→ Przykład: jak masa mózgu zmienia się w stosunku do masy ciała ?
- Czy różne klady organizmów różnią się pod względem jakiejś cechy fenotypowej ?
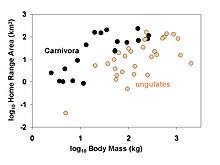
→ Przykład: czy psowate mają większe serca niż kotowate ?
- Czy grupy gatunków, które mają wspólne cechy behawioralne lub ekologiczne (np. system społeczny , dieta ) różnią się przeciętnym fenotypem?
→ Przykład: czy mięsożercy mają większy zasięg domowy niż roślinożercy?
→ Przykład: gdzie ewoluowała endotermia w linii, która doprowadziła do powstania ssaków?
→ Przykład: gdzie, kiedy i dlaczego ewoluowały łożyska i żywotność ?
- Czy cecha wykazuje znaczący sygnał filogenetyczny w określonej grupie organizmów? Czy niektóre typy cech mają tendencję do „podążania za filogenezą” bardziej niż inne?
→ Przykład: czy cechy behawioralne są bardziej niestabilne podczas ewolucji?
- Czy różnice gatunkowe w historii życia stanowią kompromis, jak w tak zwanym kontinuum szybko-wolno?
→ Przykład: dlaczego gatunki o małych ciałach żyją krócej niż ich więksi krewni?
Filogenetycznie niezależne kontrasty
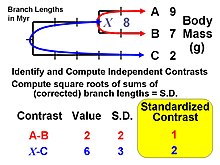
Felsenstein zaproponował w 1985 roku pierwszą ogólną metodę statystyczną do włączania informacji filogenetycznych, tj. pierwszą, która mogła wykorzystywać dowolną topologię (kolejność rozgałęzień) i określony zestaw długości rozgałęzień. Metoda jest obecnie uznawana za algorytm, który implementuje szczególny przypadek tak zwanych filogenetycznych uogólnionych modeli najmniejszych kwadratów . Logika metody jest użycie filogenicznej informacje (oraz zakładaną ruchy Browna podobnego modelu ewolucji cecha), aby przekształcić dane pierwotne końcówki (wartości średnie dla zestawu gatunków) do wartości, które są statystycznie niezależne i identycznie rozmieszczone .
Algorytm obejmuje obliczanie wartości w węzłach wewnętrznych jako krok pośredni, ale generalnie nie są one używane do samodzielnego wnioskowania . Wyjątkiem jest węzeł podstawowy (korzeniowy), który można interpretować jako oszacowanie wartości przodków dla całego drzewa (przy założeniu, że nie wystąpiły żadne kierunkowe trendy ewolucyjne [np . reguła Cope'a ]) lub jako filogenetycznie ważone oszacowanie średnia dla całego zestawu gatunków wierzchołkowych (taksony końcowe). Wartość pierwiastka jest równoważna tej otrzymanej z algorytmu „kwadratowej zmiany parsymonii” i jest również oszacowaniem największego prawdopodobieństwa w ruchu Browna. Algebry niezależnych kontrastów można również użyć do obliczenia błędu standardowego lub przedziału ufności .
Filogenetyczne uogólnione najmniejszych kwadratów (PGLS)
Prawdopodobnie najczęściej stosowanym PCM jest filogenetyczne uogólnione najmniejsze kwadraty (PGLS). To podejście służy do testowania, czy istnieje związek między dwiema (lub więcej) zmiennymi, biorąc pod uwagę fakt, że rodowód nie jest niezależny. Metoda jest szczególnym przypadkiem uogólnionych najmniejszych kwadratów (GLS) i jako taki estymator PGLS jest również bezstronny , spójny , wydajny i asymptotycznie normalny . W wielu sytuacjach statystycznych, w których stosuje się GLS (lub zwykłe najmniejszych kwadratów [OLS]) błędy resztowe ε są uważane za niezależne i identycznie rozłożone zmienne losowe, które przyjmuje się za normalne
natomiast w PGLS zakłada się rozkład błędów jako
gdzie V jest macierzą oczekiwanej wariancji i kowariancji reszt przy danym modelu ewolucyjnym i drzewie filogenetycznym. Dlatego to struktura reszt, a nie same zmienne, pokazują sygnał filogenetyczny . Od dawna jest to źródłem zamieszania w literaturze naukowej. Zaproponowano szereg modeli struktury V, takich jak ruchy Browna Ornsteina-Uhlenbecka i model λ Pagela. (Kiedy używany jest model ruchu Browna, PGLS jest identyczny z niezależnym estymatorem kontrastów). W PGLS parametry modelu ewolucyjnego są zazwyczaj współestymowane z parametrami regresji.
PGLS można zastosować tylko do pytań, w których zmienna zależna ma rozkład ciągły; jednak drzewo filogenetyczne można również włączyć do rozkładu rezydualnego uogólnionych modeli liniowych , umożliwiając uogólnienie podejścia do szerszego zestawu rozkładów odpowiedzi.
Symulacje komputerowe Monte Carlo oparte na informacjach filogenetycznych
Martins i Garland zaproponowali w 1991 roku, że jednym ze sposobów wyjaśnienia relacji filogenetycznych podczas przeprowadzania analiz statystycznych jest wykorzystanie symulacji komputerowych do stworzenia wielu zestawów danych, które są zgodne z testowaną hipotezą zerową (np. brak korelacji między dwiema cechami, brak różnicy między dwoma ekologicznie zdefiniowane grupy gatunków), ale naśladują ewolucję wzdłuż odpowiedniego drzewa filogenetycznego. Jeśli takie zestawy danych (zwykle 1000 lub więcej) są analizowane za pomocą tej samej procedury statystycznej, która jest używana do analizy rzeczywistego zestawu danych, wyniki dla zestawów danych symulowanych mogą być użyte do stworzenia filogenetycznie poprawnego (lub „PC”) rozkładów zerowych statystyka testu (np. współczynnik korelacji, t, F). Takie podejścia symulacyjne można również łączyć z takimi metodami, jak filogenetycznie niezależne kontrasty lub PGLS (patrz wyżej).
Zobacz też
- Allometria
- Ekologia behawioralna
- Bioróżnorodność
- Bioinformatyka
- Kladystyka
- Anatomia porównawcza
- Metoda porównawcza w językoznawstwie
- Fizjologia porównawcza
- Filogenetyka obliczeniowa
- Metoda pokrywania dysku
- Ekofizjologia
- Neurobiologia ewolucyjna
- Fizjologia ewolucyjna
- Uogólnione najmniejszych kwadratów (GLS)
- Uogólniony model liniowy
- Joe Felsenstein
- Mark Pagel
- Maksymalne prawdopodobieństwo
- Maksymalna oszczędność
- Paul H. Harvey
- Filogenetyka
- Strona Roderic DM
- Dobór płciowy
- Statystyka
- Systematyka
- Theodore Garland Jr.
Bibliografia
Dalsza lektura
- Ackerly, DD 1999. Ekologia porównawcza roślin i rola informacji filogenetycznej. Strony 391–413 w MC Press, JD Scholes i MG Braker, wyd. Fizjologiczna ekologia roślin. 39. sympozjum Brytyjskiego Towarzystwa Ekologicznego, które odbyło się na Uniwersytecie w Yorku 7-9 września 1998 r. Blackwell Science, Oxford, Wielka Brytania
- Berenbrink, M.; Koldkjær, P.; Kepp, O.; Cossins, AR (2005). „Ewolucja wydzielania tlenu u ryb i pojawienie się złożonego układu fizjologicznego”. Nauka . 307 (5716): 1752-1757. Kod Bib : 2005Sci...307.1752B . doi : 10.1126/science.1107793 . PMID 15774753 . S2CID 36391252 .
- Blomberg, SP; Junior ; Girlanda, T.; Ives, AR (2003). „Testowanie sygnału filogenetycznego w danych porównawczych: cechy behawioralne są bardziej labilne” (PDF) . Ewolucja . 57 (4): 717-745. doi : 10.1554/0014-3820(2003)057[0717:tfpsic]2.0.co;2 . PMID 12778543 .
- Brooks, DR i prokurator okręgowy McLennan. 1991. Filogeneza, ekologia i zachowanie: program badawczy w biologii porównawczej. Uniw. Chicago Press, Chicago. 434 s.
- Cheverud, JM; Dow, MM; Leutenegger, W. (1985). „Ilościowa ocena ograniczeń filogenetycznych w analizach porównawczych: dymorfizm płciowy w masie ciała wśród naczelnych”. Ewolucja . 39 (6): 1335-1351. doi : 10.2307/2408790 . JSTOR 2408790 . PMID 28564267 .
- Eggleton, P. i RI Vane-Wright, wyd. 1994. Filogenetyka i ekologia. Linnean Society Symposium Series Numer 17. Academic Press, Londyn.
- Felsenstein, J. 2004. Wnioskowanie filogenezy. Sinauer Associates, Sunderland, Mass. xx + 664 s.
- Freckleton, RP; Harvey, PH; Pagel, M. (2002). „Analiza filogenetyczna i dane porównawcze: test i przegląd dowodów”. Amerykański przyrodnik . 160 (6): 712–726. doi : 10.1086/343873 . PMID 18707460 . S2CID 19796539 .
- Garland Jr, T ; Ives, AR (2000). „Wykorzystywanie przeszłości do przewidywania teraźniejszości: przedziały ufności dla równań regresji w filogenetycznych metodach porównawczych” (PDF) . Amerykański przyrodnik . 155 (3): 346-364. doi : 10.1086/303327 . PMID 10718731 . S2CID 4384701 .
- Garland Jr, T.; Bennett, AF; Rezende, EL (2005). „Podejścia filogenetyczne w fizjologii porównawczej” (PDF) . Journal of Experimental Biology . 208 (16): 3015–3035. doi : 10.1242/jeb.01745 . PMID 16081601 . S2CID 14871059 .
- Garland Jr, T.; Harvey, PH; Ives, AR (1992). „Procedury analizy danych porównawczych z wykorzystaniem filogenetycznie niezależnych kontrastów” (PDF) . Biologia systematyczna . 41 (1): 18–32. doi : 10.2307/2992503 . JSTOR 2992503 .
- Gittleman, JL; Kot, M. (1990). „Adaptacja: statystyki i model zerowy do szacowania efektów filogenetycznych”. Zoologia systematyczna . 39 (3): 227–241. doi : 10.2307/2992183 . JSTOR 2992183 .
- Hadfield, JD; Nakagawa, S. (2010). „Ogólne ilościowe metody genetyczne biologii porównawczej: filogenezy, taksonomie i modele wielocechowe dla znaków ciągłych i kategorycznych” . Czasopismo Biologii Ewolucyjnej . 23 (3): 494–508. doi : 10.1111/j.1420-9101.2009.01915.x . PMID 20070460 . S2CID 27706318 .
-
Herrada, EA; Tesson, CJ; Klemm, K.; Eguiluz, VM; Hernandez-Garcia, E.; Duarte CM (2008). „Powszechne skalowanie w rozgałęzianiu drzewa życia” . PLOS 1 . 3 (7): e2757. arXiv : 0807.4042 . Kod Bib : 2008PLoSO....3.2757H . doi : 10.1371/journal.pone.0002757 . PMC 2447175 . PMID 18648500 .

- Housworth, EA; Martins, PE; Lynch, M. (2004). „Filogenetyczny model mieszany” (PDF) . Amerykański przyrodnik . 163 (1): 84–96. doi : 10.1086/380570 . PMID 14767838 . S2CID 10568814 .
- Ives, AR 2018. Modele mieszane i filogenetyczne: koncepcyjne wprowadzenie do skorelowanych danych. leanpub.com, 125 s., https://leanpub.com/correlateddata
- Ives, AR; Midford, PE; Garland Jr, T. (2007). „Zmienność wewnątrzgatunkowa i błąd pomiaru w filogenetycznych metodach porównawczych” . Biologia systematyczna . 56 (2): 252–270. doi : 10.1080/1063515070701313830 . PMID 17464881 .
- Maddison, DR (1994). „Filogenetyczne metody wnioskowania o historii ewolucyjnej i procesie zmian dyskretnie cenionych postaci”. Roczny Przegląd Entomologii . 39 : 267–292. doi : 10.1146/annurev.ento.39.1.267 .
- Maddison, WP (1990). „Metoda testowania skorelowanej ewolucji dwóch postaci binarnych: Czy zyski lub straty są skoncentrowane na niektórych gałęziach drzewa filogenetycznego?”. Ewolucja . 44 (3): 539–557. doi : 10.2307/2409434 . JSTOR 2409434 . PMID 28567979 .
- Maddison, WP i DR Maddison. 1992. MacClade. Analiza filogenezy i ewolucji charakteru. Wersja 3. Sinauer Associates, Sunderland, Mass. 398 s.
- Martins, EP, wyd. 1996. Filogenezy i metoda porównawcza w zachowaniu zwierząt. Oxford University Press, Oksford. 415 stron.
- Martins, PE; Hansen, TF (1997). „Phylogenies i metoda porównawcza: ogólne podejście do włączania informacji filogenetycznych do analizy danych międzygatunkowych” . Amerykański przyrodnik . 149 (4): 646–667. doi : 10.1086/286013 . S2CID 29362369 .Errata Am. Nat. 153:448.
- Nunn, CL; Barton, RA (2001). „Porównawcze metody badania adaptacji i allometrii naczelnych” . Antropologia ewolucyjna . 10 (3): 81–98. doi : 10.1002/evan.1019 . S2CID 16959643 .
- Oakley, TH; Gu, Z.; Abouheif, E.; Patel, NH; Li, W.-H. (2005). „Metody porównawcze analizy ewolucji ekspresji genów: przykład wykorzystujący funkcjonalne dane genomowe drożdży” (PDF) . Biologia molekularna i ewolucja . 22 : 40–50. doi : 10.1093/molbev/msh257 . PMID 15356281 .
- O'Meara, BC; Ane CM; Sandersona, MJ; Wainwright, PC (2006). „Testowanie różnych szybkości ciągłej ewolucji cech w różnych grupach przy użyciu prawdopodobieństwa” (PDF) . Ewolucja . 60 : 922–933. doi : 10.1554/05-130.1 . S2CID 13796463 .
- Organ, CL; Shedlock, AM; Meade, A.; Pagel, M.; Edwards, SV (2007). „Pochodzenie wielkości i struktury ptasiego genomu u nie ptasich dinozaurów”. Natura . 446 (7132): 180–184. Kod Bibcode : 2007Natur.446..180O . doi : 10.1038/nature05621 . PMID 17344851 . S2CID 3031794 .
- Strona, RDM , wyd. 2003. Drzewa splątane: filogeneza, kospecjacja i koewolucja. University of Chicago Press, Chicago.
- Pagel, MD (1993). „W poszukiwaniu ewolucyjnego współczynnika regresji: analiza tego, co mierzą metody porównawcze”. Czasopismo Biologii Teoretycznej . 164 (2): 191-205. doi : 10.1006/jtbi.1993.1148 . PMID 8246516 .
- Pagel, M (1999). „Wnioskowanie historycznych wzorców ewolucji biologicznej”. Natura . 401 (6756): 877-884. Kod Bib : 1999Natur.401..877P . doi : 10.1038/44766 . hdl : 2027,42/148253 . PMID 10553904 . S2CID 205034365 .
- Paradis, E (2005). „Analiza statystyczna zróżnicowania cechami gatunkowymi” (PDF) . Ewolucja . 59 (1): 1–12. doi : 10.1554/04-231 . PMID 15792222 . S2CID 196612333 .
- Raj.; Claude, J. (2002). „Analiza danych porównawczych z wykorzystaniem uogólnionych równań estymujących” (PDF) . Czasopismo Biologii Teoretycznej . 218 (2): 175–185. doi : 10.1006/jtbi.2002.3066 . PMID 12381290 .
- Purvis, A.; Garland Jr, T. (1993). „Polytomie w analizach porównawczych znaków ciągłych” (PDF) . Biologia systematyczna . 42 (4): 569–575. doi : 10.2307/2992489 . JSTOR 2992489 .
- Rezende, EL i T. Garland, Jr. 2003. Comparaciones interespecíficas y métodos estadísticos filogenéticos. Strony 79–98 w F. Bozinovic, wyd. Fisiología Ecológica & Evolutiva. Teoría y casos de estudios en animales. Ediciones Universidad Católica de Chile, Santiago. PDF
- Rezende, El; Diniz-Filho, JAF (2012). „Analizy filogenetyczne: porównywanie gatunków w celu wywnioskowania adaptacji i mechanizmów fizjologicznych” (PDF) . Kompleksowa Fizjologia . 2 (1): 639–674. doi : 10.1002/cphy.c100079 . PMID 23728983 .
- Ridley, M. 1983. Wyjaśnienie różnorodności organicznej: metoda porównawcza i adaptacje do krycia. Clarendon, Oksford, Wielka Brytania
- Rohlf, FJ (2001). „Porównawcze metody analizy zmiennych ciągłych: interpretacje geometryczne” . Ewolucja . 55 (11): 2143–2160. doi : 10.1111/j.0014-3820.2001.tb00731.x . PMID 11794776 . S2CID 23200090 .
- Rohlf, FJ (2006). „Komentarz do korekty filogenetycznej”. Ewolucja . 60 (7): 1509–1515. doi : 10.1554/05-550.1 . PMID 16929667 . S2CID 198156300 .
- Sanford, dyrektor generalny; Lutterschmidt, WI; Hutchison, VH (2002). „Ponowna metoda porównawcza” . Bionauka . 52 (9): 830-836. doi : 10.1641/0006-3568(2002)052[0830:tcmr]2.0.co;2 .
- Schluter, D.; Cena, T.; Mooers, AO; Ludwig, D. (1997). „Prawdopodobieństwo stanów przodków w promieniowaniu adaptacyjnym”. Ewolucja . 51 (6): 1699-1711. doi : 10.2307/2410994 . JSTOR 2410994 . PMID 28565128 .
- Smith, RJ; Cheverud, JM (2002). „Skalowanie dymorfizmu rozmiaru płciowego w masie ciała: analiza filogenetyczna reguły Renscha u naczelnych”. Międzynarodowy Czasopismo Prymatologiczne . 23 (5): 1095-1135. doi : 10.1023/A:1019654100876 . S2CID 42439809 .
- Steppan, SJ; Phillips, PC; Houle, D. (2002). „Porównawcza genetyka ilościowa: ewolucja macierzy G” (PDF) . Trendy w ekologii i ewolucji . 17 (7): 320–327. doi : 10.1016/s0169-5347(02)02505-3 .
- Vanhooydonck, B.; Van Damme, R. (1999). „Ewolucyjne relacje między kształtem ciała a wykorzystaniem siedlisk u jaszczurek lacertid”. Badania Ekologii Ewolucyjnej . 1 : 785–805.
Zewnętrzne linki
- Wykład online z adaptacji i metody porównawczej, z praktycznym przykładem filogenetycznie niezależnych kontrastów i quizem mistrzowskim
- Lista programów filogenetycznych
- Phylocomm
- Narzędzia filogenetyczne dla biologii porównawczej
- Strona internetowa Filogeneza Snu
- Drzewo życia
Czasopisma
- Amerykański przyrodnik
- Ekologia behawioralna
- Ekologia
- Ewolucja
- Badania ekologii ewolucyjnej
- Funkcjonalna Ekologia
- Journal of Evolutionary Biology
- Transakcje filozoficzne Royal Society of London B
- Zoologia fizjologiczna i biochemiczna
- Biologia systematyczna
Pakiety oprogramowania (lista niepełna)
- Analizy Filogenetyki i Ewolucji
- BayesCechy
- Analiza porównawcza przez niezależne kontrasty
- PORÓWNYWAĆ
- Lista Felsensteina
- Mesquite PDAP: PDTree dla Mesquite
- mvMorph
- ouch: Ornstein-Uhlenbeck dla hipotez porównawczych
- PDAP: programy analizy różnorodności fenotypowej
- Phylocom
- Regresja filogenetyczna
- FIZYKA